查看更多

 明星产品
明星产品
 关注我们
关注我们

扫码关注我们
了解更多信息
在前面几期内容中,已经给大家介绍了CRISPR/cas9在基因编辑中的应用与基因编辑的详细操作步骤,但是CRISPR/cas9对基因组进行编辑时会使基因组上发生片段插入或缺失,改变基因组本身结构,因此改造CRISPR/cas9工具,使其在转录或转录后水平上调控基因表达成为其应用拓展的关键。科学家们通过对cas9蛋白进行改造后,进一步拓展了其应用范围,今天,小恒就带大家一起来了解下cas9系统在转录调控中的应用。

一、dCas9调控基因转录原理
Cas9蛋白具有独立结合DNA和切割DNA的结构域,对DNA的切割主要依赖于HNH和RuvC结构域,这两个结构域相互独立,分别去切割DNA的两条链,将HNH和RuvC的切割位点突变后既不影响蛋白质整体结构,同时又可以使其失去对DNA的切割能力,突变后的cas9蛋白可以在sgRNA的引导下结合到DNA上,但不对DNA起切割作用。这时候如果在cas9蛋白的另一端融合适当的效应器,就可以实现对靶基因进行表观修饰或直接作用于其表达调控元件从而对内源基因进行表达调控(Dominguez AA et al.,2016)。这种HNH和RuvC突变的cas9蛋白,我们称其为dcas9(dead-cas9),根据融合的不同效应其可将其大致分为CRISPRi(CRISPR interference0029和CRISPRa(CRISPR activation).
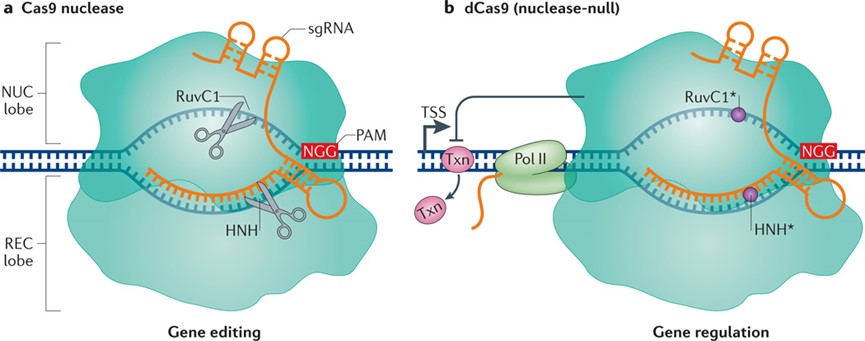
图1.Cas9及dCas9工作原理图:A,cas9基因编辑;B,dcas9调控基因表达(Dominguez AA et al.,2016)
二、CRISPRi:dcas9与基因抑制
CRISPRi即将抑制基因转录的效应器融合到cas9蛋白上,在sgRNA的引导下,转录抑制效应蛋白与靶基因相关元件作用抑制靶基因的转录表达,目前常用的CRISPRi工具有:dCas9-KRAB,dCas9-DNMT3A,dCas9-HDAC和CRISPRoff。
1、dCas9-KRAB
编码基因中的KruppelAssociation Boxes(KRAB)结构域具有转录抑制作用,人类基因组中大约有350个编码蛋白的基因含有该结构域。Gilbert将dCas9蛋白与KRAB结构域融合,构建dCas9-KRAB融合蛋白,该融合蛋白可以在sgRNA的指导下特异性地靶向EGFP载体以降低荧光活性(Gilbert Luke A et al.,2013)。研究表明dCas9-KRAB通过将甲基转移酶SETDB1招募到靶点来对靶基因进行甲基化修饰从而抑制靶基因的表达。dcas9-KRAB还可以靶向基因调控元件,例如,dCas9-KRAB可以靶向HS2增强子,增加增强子H3K9me3的修饰,减少增强子和启动子染色体的可及性,并沉默多个珠蛋白基因的表达。虽然该平台融合了KOX1的KRAB结构域,在大多数情况下可以特异性地抑制靶基因的表达,但在某些情况下,它不能完全沉默基因的表达,这可能会影响后续的研究。相比之下,ZIM3-KRAB更能有效地抑制基因表达。目前,dCas9-KRAB融合蛋白是用于基因抑制、基因功能研究、基因调控元件筛选和疾病治疗筛选的最广泛的工具。
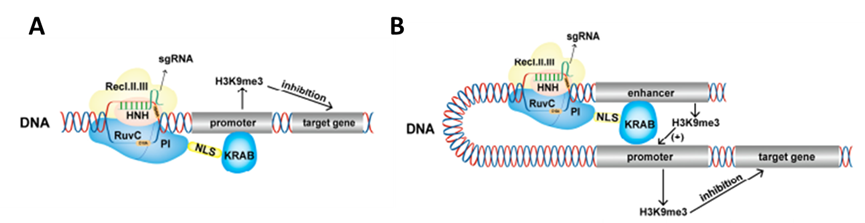
图2.dcas9-krab抑制基因表达原理:A,dcas9-krab调控启动子;B, dcas9-krab调控增强子(Cai et al.,2023)
2、dCas9-DNMT3A
DNA甲基化转移酶(DNMT)可以催化CpG岛DNA甲基化,从而抑制基因表达。Vojta等人(Vojta et al., 2016)将dCas9蛋白连接到DNMT3A的催化域,开发了一种dCas9-DNMT3A融合蛋白,该融合蛋白可直接导致IL6ST和BACH基因启动子区域的DNA甲基化,抑制基因表达。Liu等人(Liu et al.,2016)使用dCas9-DNMT3A融合蛋白对CTCF环进行靶向甲基化,阻断CTCF结合并干扰DNA循环,从而改变相邻环中的基因表达。dCas9-DNMT3A融合蛋白可以靶向EpCAM、CXCR4、TFRC和其他基因的启动子区域,并导致靶点CpG岛的甲基化,导致靶基因的转录抑制。然而,dCas9-DNMT3A融合蛋白的非靶向效应限制了DNA甲基化的评估, Pflueger等人(Pflueger et al.,2018)将Suntag阵列融合到dCas9蛋白中,构建了dCAS9Sun-D3A系统,该系统使用dCas9-Suntag将DNMT3A蛋白质招募到靶点,这可以独立调节DNMT3A催化域和dCas9Sun-Tag的表达,实现更准确和高效的DNA甲基化编辑。
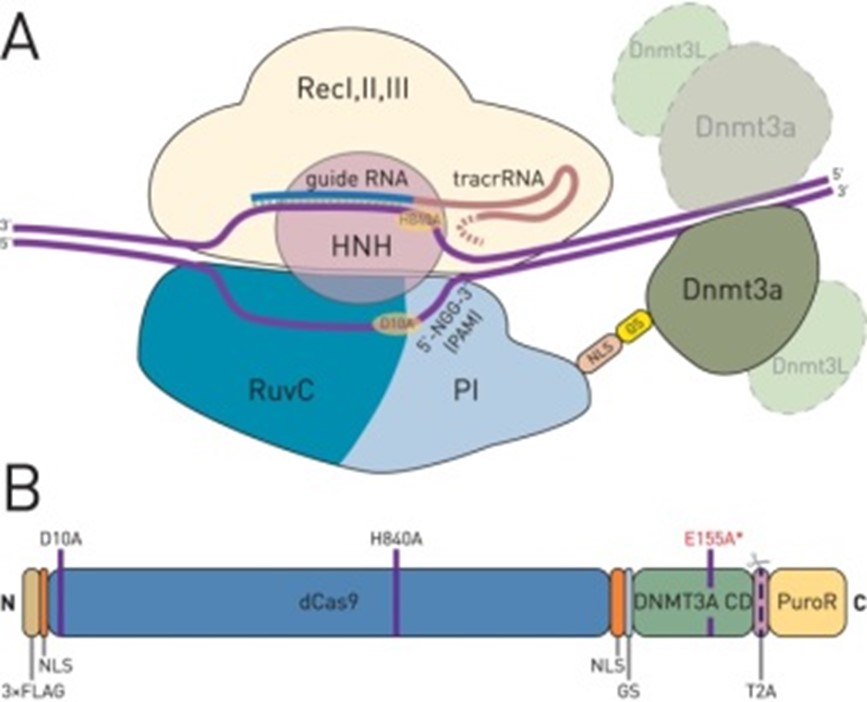
图3.dcas9- DNMT3A抑制基因表达 (Vojta Aleksandar et al.,2016)
3、 dCas9-HDAC
组蛋白去乙酰酶(HDAC)在体内主要催化去乙酰化过程,可通过降低基因调控区组蛋白乙酰化水平来抑制靶基因的表达。Chen等人[Chen et al.,2019]使用dCas9-HDAC8融合蛋白来阻断Fos基因增强子的组蛋白乙酰化,从而抑制Fos的表达,从而显著降低神经元ON的比例,延长启动子处于OFF状态的时间,并减少总的猝发频率。活性诱导组蛋白去乙酰化可以调节基因转录,从而影响神经元的基因表达和细胞功能。与其他CRISPR工具一样,dCas9-HDAC可以诱导基因组脱乙酰化,用于研究表观遗传修饰对生物过程的影响
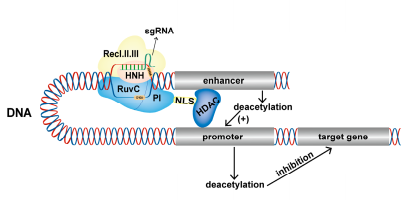
图4.dCas9-HDAC抑制基因表达原理(Cai et al.,2023)
4、 CRISPRoff
CRISPRi工具在短时间内可以调节目标基因的表达,并且不是可遗传。Nunez等人(Nunez et al.,2021)将DNMT3A、DNMT3L和KRAb融合到dCas9蛋白上,构建了CRISPRoff结构(DNMT3A-DNMT3L-dCas9-KRAb)。瞬时表达CRISPRoff导致高度特异的DNA甲基化,并通过干细胞的分裂和分化维持基因抑制。此外,CRISPRoff的表观遗传沉默不仅限于具有典型CpG岛的基因,而且可以沉默没有CpG岛结构的基因。此外,由于它针对不同基因的许多启动子和增强子,CRISPRoff在基因组中表现出广泛而稳定的基因沉默,表明CRISPRoff可以诱导稳定的表观遗传记忆。提供了比其他CRISPRi工具更多的选项来研究基因甲基化修改对基因功能和灵活的基因表达调控的影响。
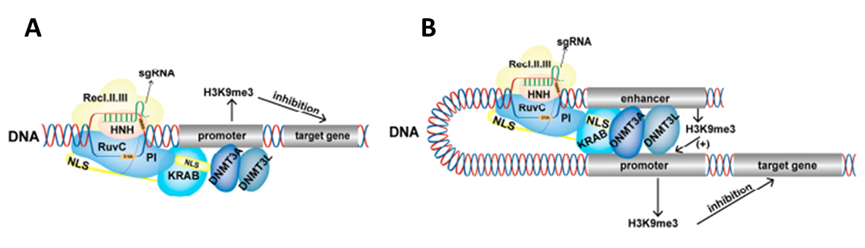
图5.CRISPRoff抑制基因表达原理:A, CRISPRoff调控启动子;B,CRISPRoff调控增强子(Cai et al.,2023)
三、CRISPRa :dcas9与基因激活。
CRISPRa即将激活基因转录的效应器融合到cas9蛋白上,在sgRNA的引导下,转录激活效应蛋白与靶基因相关元件作用激活靶基因的转录表达,目前常用的CRISPRa工具有:dCas9-VP64,dCas9-VPR,dCas9-p300和dCas9-dMSK1。
1、 dCas9-VP64
VP64是单纯疱疹病毒16(VP16)的四聚体,可在转录水平激活基因表达。Gilbert等人(Gilbert Luke A et al.,2013)将dCas9与VP64融合蛋白,有效地激活了Gal4UAS-GFP的表达。此外,在特异靶向NTF3和VEGFA的单个或多个sgRNA的引导下,dCas9-VP64显著增加了基因的表达,表明该融合蛋白可以特异性地激活内源性人类基因的表达。dCas9-VP64是一种有效的CRISPR激活工具,已被广泛用于研究多基因的激活和一些疾病治疗。
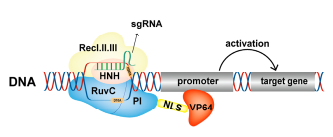
图6.dCas9-VP64激活基因表达原理(Cai et al.,2023)
2、 dCas9-VPR
P65是核因子-κB家族的成员,是一种转录因子,具有REL样结构域,可激活靶基因的表达。Gilbert等(Gilbert Luke A et al.,2013)构建了dCas9-p65融合蛋白,证实其能有效激活靶基因。由于dCas9-p65融合蛋白含有单一的p65激活结构域,因此它对靶基因的激活作用有限。为了提高其效果,Lin等人将高活性的VP64-P65-RTA结构域与dCas9蛋白融合,构建了dCas9-VPR激活系统(Lin, S.et al.,2015)。在果蝇细胞和体内研究的特性分析表明,dCas9-VPR能有效地激活靶点,并导致体内的优势表型。dCas9-VPR系统已经被用于上调基因表达,来研究基因的功能。除了特定的激活蛋白编码基因外,dCas9-VPR系统还可以有效地靶向lncRNA的表达。为了探讨lncRNA DANCR在大鼠脂肪干细胞软骨形成和骨愈合特性中的作用,Nuong Thi Kieu等人(Nguyen et al.,2021)通过dCas9-VPR特异性地上调了内源性lncRNA DANCR的表达。激活DANCR的表达显著促进了大鼠颅骨的愈合,为促进颅骨愈合提供了潜在的治疗策略。
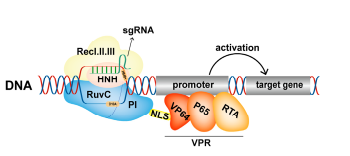
图7.dCas9-VPR激活基因表达原理(Cai et al.,2023)
3、dCas9-p300
P300蛋白是一种重要的组蛋白乙酰转移酶,可以激活基因表达,参与多种生物学过程。Hilton等人(Hilton, et al.,2015)设计了三种融合蛋白:p300全长序列(DCas9FLp300)、p300乙酰基转移酶(HAT)活性核心区(dCas9p300-core(WT))和p300乙酰基转移酶核(HAT)活性核心区突变体(dCas9p300-core(D1399Y))。 WT融合蛋白促进目标基因启动子和远端调控元件的组蛋白乙酰化,并更有效和特异地激活目标基因的表达。与其他CRISPRa转录激活工具相比,dCas9p300-core(WT)具有更好的内在配体调节能力,为未来特定基因组位点的时空乙酰化调控奠定了基础。
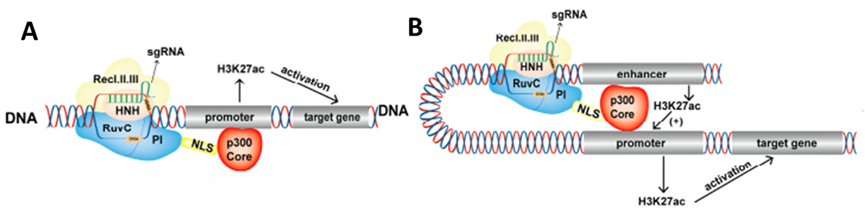
图8.dCas9-p300激活基因表达原理:A, dCas9-p300调控启动子;B,dCas9-p300调控增强子(Cai et al.,2023)
4、dCas9-dMSK1
丝裂原和应激激活蛋白激酶1(MSK1)催化组蛋白H3S10和H3S28的磷酸化。Li等(Li et al.,2022)利用MSK1在催化磷酸化中的乙酰化活性设计了三种融合蛋白:连接MSK1全长的dCas9-MSK1,连接MSK1催化核心区的dCas9-dMSK1,以及包括dMSK1突变的dCas9-ddMSK1。DCas9-dMSK1融合蛋白具有更好的催化活性,导致组蛋白H3S28在靶位的磷酸化水平提高。CHIP-SEQ和RNA-SEQ结果表明,dCas9-MSK1能特异性结合和催化OCT4基因启动子上组蛋白H3S28的磷酸化,并有效地诱导其表达。dCas9-dMSK1是研究组蛋白磷酸化和靶向激活基因的有力工具,扩展了这种表观遗传编辑工具的功能。
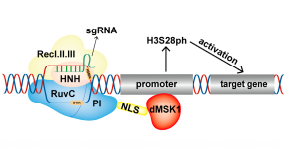
图9.dCas9-dMSK1激活基因表达原理(Cai et al.,2023)
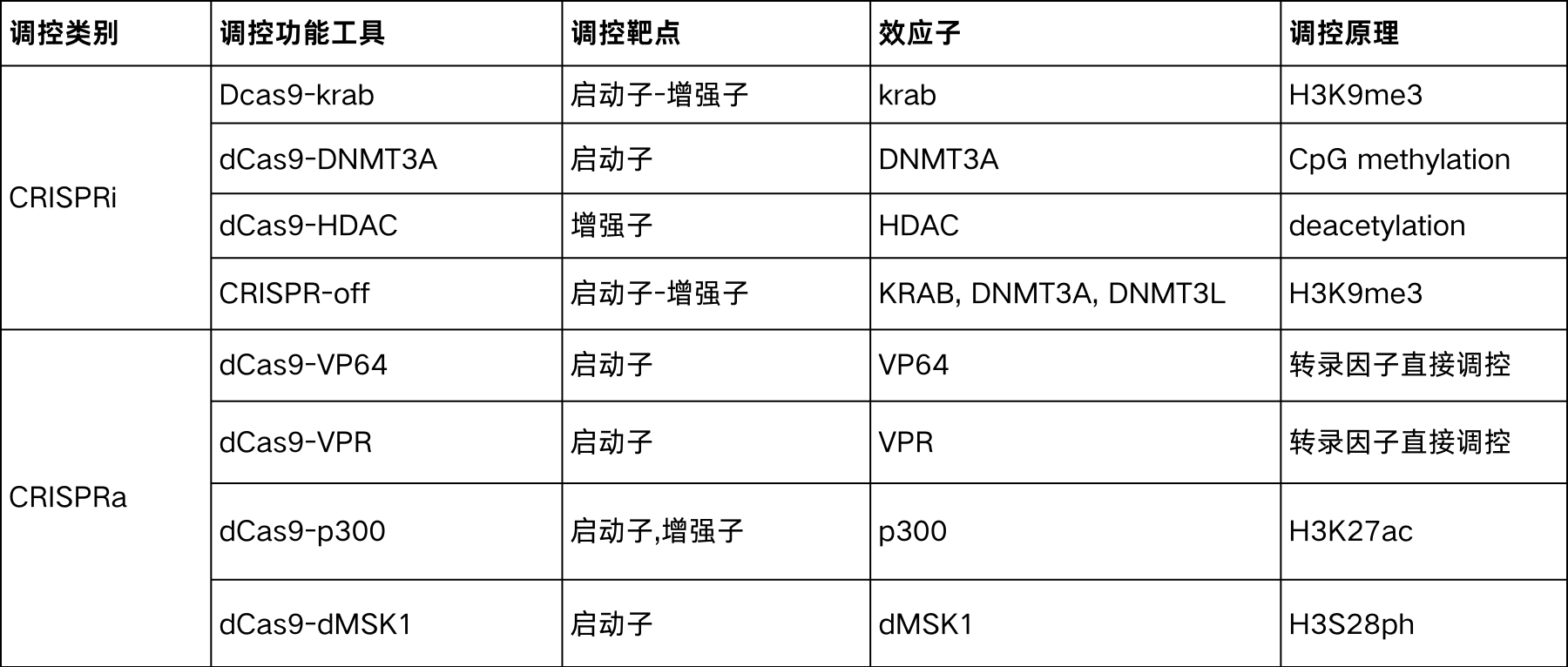
dcas9对基因的内源表达调控主要是利用了cas9 DNA结合域和具有切割酶活性的结构域在结构和功能上的相对独立的特征,将可以调节基因转录的转录调控因子或表观修饰基因作为效应子与dcas9蛋白融合,利用sgRNA引导dcas9-effector共同作用于靶基因启动子部分,调控基因表达。这些效应子有的作为转录因子调控基因的表达调控,有的作为表观修饰蛋白改变启动子或增强子元件的表观修饰水平从而改变基因的转录活性,实现在不改变基因组结构的情况下实现对内源基因的表达调控。
表1.不同dcas9工具调控内源基因表达的方式
针对一些通过常规过表达载体难以实现过表达或者难以设计干扰序列的基因,利用dcas9工具直接调控细胞内源表达是一个不错的选择。利用dcas9工具进行内源基因表达调控与cas9基因编辑流程基本一致,可以参考本系列的基因敲除篇中sgRNA设计一文(前文链接),与基因编辑不同的是,dcas9基因调控的sgRNA需要设计在启动子区域,验证sgRNA的活性后,将sgRNA与相应的dcas9工具共转靶细胞,并检测靶基因的表达变化。
汉恒生物有dcas9-krab、dcas9-DNMT3A、dcas9-vpr、dcas9-vp64等基因表达调控腺病毒系统,若有相关需求,欢迎咨询汉恒生物微信公众号或拨打官网技术服务热线~
四、参考文献
1.Dominguez Antonia A,Lim Wendell A,Qi Lei S,Beyond editing: repurposing CRISPR-Cas9 for precision genome regulation and interrogation.[J] .Nat Rev Mol Cell Biol, 2016, 17: 5-15.
2.Gilbert Luke A,Larson Matthew H,Morsut Leonardo et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes.[J] .Cell, 2013, 154: 442-51.
3.2Cai Ruijie,Lv Runyu,Shi Xin'e et al. CRISPR/dCas9 Tools: Epigenetic Mechanism and Application in Gene Transcriptional Regulation.[J] .Int J Mol Sci, 2023, 24: undefined.
4.DNMAT3 Vojta Aleksandar,Dobrinić Paula,Tadić Vanja et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation.[J] .Nucleic Acids Res, 2016, 44: 5615-28.
5. Vojta, A.; Dobrinic, P.; Tadic, V.; Bockor, L.; Korac, P.; Julg, B.; Klasic, M.; Zoldos, V. Repurposing the CRISPR-Cas9 system fortargeted DNA methylation. Nucleic Acids Res. 2016, 44, 5615–5628
6.Chen, L.F.; Lin, Y.T.; Gallegos, D.A.; Hazlett, M.F.; Gomez-Schiavon, M.; Yang, M.G.; Kalmeta, B.; Zhou, A.S.; Holtzman, L.;Gersbach, C.A.; et al. Enhancer Histone Acetylation Modulates Transcriptional Bursting Dynamics of Neuronal Activity-InducibleGenes. Cell Rep. 2019, 26, 1174–1188.e1175.
7. Liu, X.S.; Wu, H.; Ji, X.; Stelzer, Y.; Wu, X.; Czauderna, S.; Shu, J.; Dadon, D.; Young, R.A.; Jaenisch, R. Editing DNA Methylationin the Mammalian Genome. Cell 2016, 167, 233–247.e217.
8. Pflueger, C.; Tan, D.; Swain, T.; Nguyen, T.; Pflueger, J.; Nefzger, C.; Polo, J.M.; Ford, E.; Lister, R. A modular dCas9-SunTagDNMT3A epigenome editing system overcomes pervasive off-target activity of direct fusion dCas9-DNMT3A constructs. Genome Res. 2018, 28, 1193–1206.
9.Nunez, J.K.; Chen, J.; Pommier, G.C.; Cogan, J.Z.; Replogle, J.M.; Adriaens, C.; Ramadoss, G.N.; Shi, Q.; Hung, K.L.; Samelson, A.J.;et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell 2021, 184, 2503–2519.e2517.
10.Lin, S.; Ewen-Campen, B.; Ni, X.; Housden, B.E.; Perrimon, N. In vivo transcriptional activation using CRISPR-Cas9 in Drosophila.Genetics. 2015, 201, 433–442.
11.Nguyen, N.T.K.; Chang, Y.H.; Truong, V.A.; Hsu, M.N.; Pham, N.N.; Chang, C.W.; Wu, Y.H.; Chang, Y.H.; Li, H.; Hu, Y.C. CRISPRactivation of long non-coding RNA DANCR promotes bone regeneration. Biomaterials 2021, 275, 120965.
12.Hilton, I.B.; D’Ippolito, A.M.; Vockley, C.M.; Thakore, P.I.; Crawford, G.E.; Reddy, T.E.; Gersbach, C.A. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat. Biotechnol. 2015, 33, 510–517.
13. Li, J.; Mahata, B.; Escobar, M.; Goell, J.; Wang, K.; Khemka, P.; Hilton, I.B. Programmable human histone phosphorylation and gene activation using a CRISPR/Cas9-based chromatin kinase. Nat. Commun. 2021, 12, 896.

查看更多

查看更多

查看更多

联系我们

返回顶部