查看更多

 明星产品
明星产品
 关注我们
关注我们

扫码关注我们
了解更多信息
在1998年Andrew Fire 和Craig Mello发现了一种双链RNA,这种RNA能抑制秀丽隐杆线虫基因的表达,他们将这种现象命名为RNA干扰(RNA interference,RNAi)1。从发现至今,RNAi 是科研中最常见的基因缺失策略,具有高度的特异性,可用于调控基因和蛋白质的表达,这是传统的药理学方法难以企及的2。

RNAi 可以通过两种分子来实现:一种是通过化学合成的双链小干扰 RNAinterfering RNA ,siRNA);另一种是构建到载体上的短发夹RNA(short hairpin RNA ,shRNA)。siRNA 是最早应用在RNAi上的,随后人们发现通过将siRNA添加到一个短发夹结构上并构建到质粒上,能被细胞的RNA聚合酶III识别并启始转录1,3,4。虽然siRNA和shRNA有相同的功能,但是分子作用机制、干扰途径、脱靶效率和应用范围是有所不同的,本质上是两种不同的分子5。
siRNA
siRNA 是人工直接合成的,一般是21-25nt的带有两个游离双链RNA,通过转染试剂(如RNAFit)将 siRNA 转入到细胞的细胞质中,与细胞质中的一些蛋白相互作用,其中的一条链与一些蛋白形成转录沉默复合物5。
siRNA 适用条件
siRNA 是人工合成的,相对价格较为便宜,虽然对 siRNA 的效果的预测方法已经日趋成熟,但是siRNA效果仍需要试验去验证,所以在经济条件允许的情况下建议多定制几条siRNA去筛选出一条干扰效果最好 siRNA 也不失为一种节省时间的好方法。此外值得注意的是siRNA 的转染是需要转染试剂的,如果要研究的细胞对转染试剂敏感,容易死亡,需要更换其他的转染试剂或者改用病毒携带shRNA。

此外,siRNA可以通过化学修饰添加不同的功能,比如汉恒的siRNA产品中会带有荧光标记的阴性对照(如Cy3),转染后3h左右,能在荧光显微镜下看到荧光6,通过这个方法可以判断siRNA是否能进入到细胞内,以此排除转染过程中是否存在问题。此外,已有大量的研究通过化学修饰,来增加siRNA的稳定性和细胞的亲和力,同时也要兼顾对干扰功能的影响7,比如在动物实验中常用到的甲氧基和胆固醇双修饰。
shRNA
shRNA包含一个正义序列和一个反义序列,并由环结构分隔。与siRNA不同的是,shRNA需要在细胞内转录生成的,在细胞核和细胞质中加工后才形成具有沉默作用的 RISC,为了提高shRNA的加工效率,这一个过程被设计的有点类似miRNA的成熟过程5,此外使用非U6 启动子时,要把shRNA上添加miR-30结构8。
因为shRNA是通过载体(质粒或病毒)转录而来,这样shRNA就具备siRNA不能具有的优势:
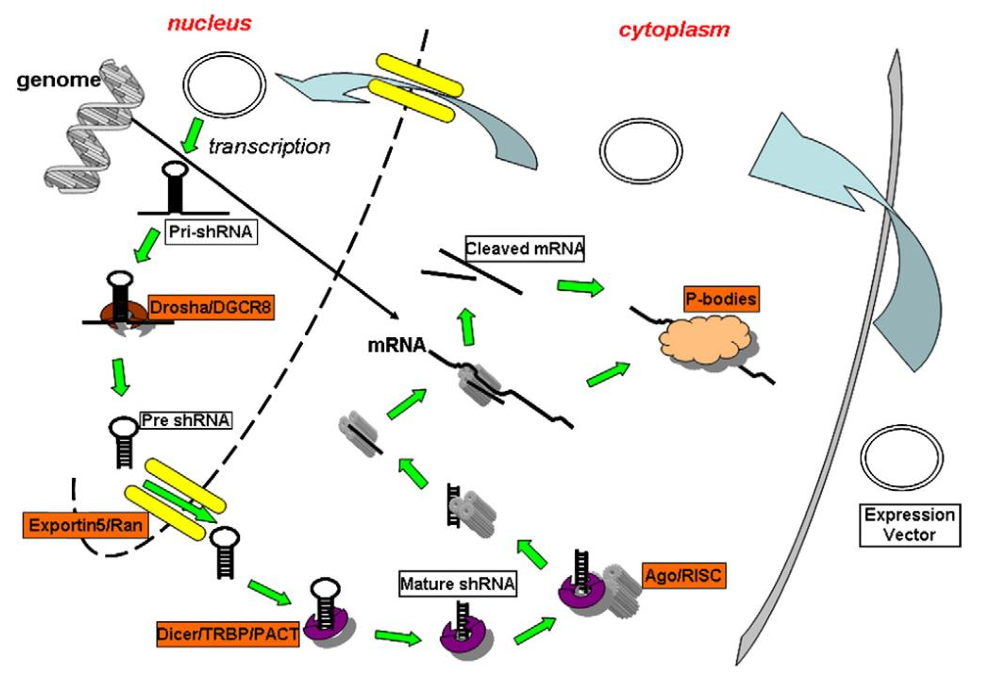
综上,选择siRNA还是shRNA要根据自己的实际需求而定的,两种都是可行的方法。
1. Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 1998;391(6669):806-811.
2. Liu S. RNA Interference Ex Vivo. Methods in molecular biology (Clifton, NJ). 2018;1868:129-135.
3. Yu JY, DeRuiter SL, Turner DL. RNA interference by expression of short-interfering RNAs and hairpin RNAs in mammalian cells. Proceedings of the National Academy of Sciences of the United States of America. 2002;99(9):6047-6052.
4.Miyagishi M, Taira K. U6 promoter-driven siRNAs with four uridine 3' overhangs efficiently suppress targeted gene expression in mammalian cells. Nature biotechnology. 2002;20(5):497-500.
5. Rao DD, Vorhies JS, Senzer N, Nemunaitis J. siRNA vs. shRNA: similarities and differences. Advanced drug delivery reviews. 2009;61(9):746-759.
6. Järve A, Müller J, Kim IH, et al. Surveillance of siRNA integrity by FRET imaging. Nucleic acids research. 2007;35(18):e124.
7. Takabatake Y, Isaka Y, Mizui M, Kawachi H, Takahara S, Imai E. Chemically modified siRNA prolonged RNA interference in renal disease. Biochemical and biophysical research communications.2007;363(2):432-437.
8. Silva JM, Li MZ, Chang K, et al. Second-generation shRNA libraries covering the mouse and human genomes. Nature genetics. 2005;37(11):1281-1288.
9. Kühn R, Streif S, Wurst W. RNA interference in mice. Handbook of experimental pharmacology. 2007(178):149-176.

查看更多

查看更多

查看更多

联系我们

返回顶部