查看更多

 明星产品
明星产品
 关注我们
关注我们

扫码关注我们
了解更多信息
上期内容系统介绍了组蛋白甲基化在基因表达调控中的作用,本期我们继续为大家介绍组蛋白的乙酰化修饰。

什么是组蛋白乙酰化?
组蛋白乙酰化作为另一种重要的表观遗传修饰,同样通过动态可逆的化学修饰参与染色质结构的调控,在转录起始和延伸、基因沉默和表观遗传细胞记忆中起着重要的调节作用。与甲基化类似,组蛋白乙酰化过程也受到writers(HAT,组蛋白乙酰转移酶)、erasers(HDAC,组蛋白脱乙酰酶)和readers(BRD,溴结构域)三类蛋白的精密调控。研究表明,在多种疾病尤其是肿瘤进展过程中,HATs、HDACs及BRD的功能异常或表达失调可导致表观遗传紊乱,进而影响关键基因的转录过程。因此,这些调控因子已成为极具潜力的表观遗传治疗靶点。
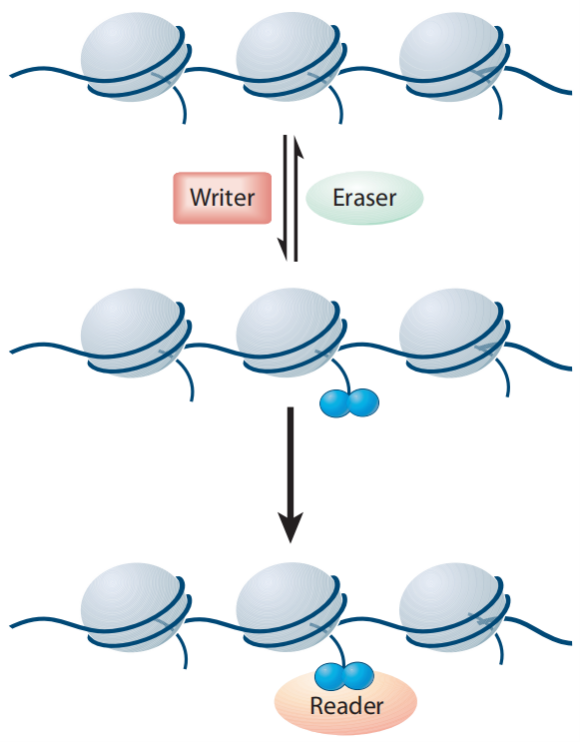
图1、组蛋白翻译后修饰的writing、erasing和reading示意图[1]
组蛋白乙酰化的分子机制
从分子机制上看,组蛋白乙酰化水平的动态平衡直接依赖于组蛋白乙酰转移酶(histone Acetyl Transferases, HAT)和组蛋白脱乙酰酶(histone deacetylases, HDAC)的协同作用。HATs通过将乙酰基从乙酰辅酶A转移到组蛋白尾巴上的赖氨酸(Lys或K)残基基团上来实现组蛋白的乙酰化,这一过程导致组蛋白上的正电荷被中和,进而削弱了组蛋白与DNA(带负电荷的磷酸基团)之间的相互作用,因此,染色质结构变得分散,不再紧凑,从而更容易被转录因子接近[2, 3]。而逆反应中,HDACs去除高乙酰化组蛋白上的乙酰基,从而导致低水平的乙酰化、常染色质向异染色质转变以及基因沉默[4]。值得注意的是,HATs和HDACs的调控范围并不限于组蛋白,它们同样修饰非组蛋白,所以一些团队也建议将HAT和HDAC重命名为赖氨酸乙酰转移酶(KAT)或赖氨酸脱乙酰酶(KDAC)以更准确反映其功能[5]。鉴于本文聚焦于表观遗传调控机制,下文仍沿用传统命名,以方便阐述其对组蛋白乙酰化的核心作用。
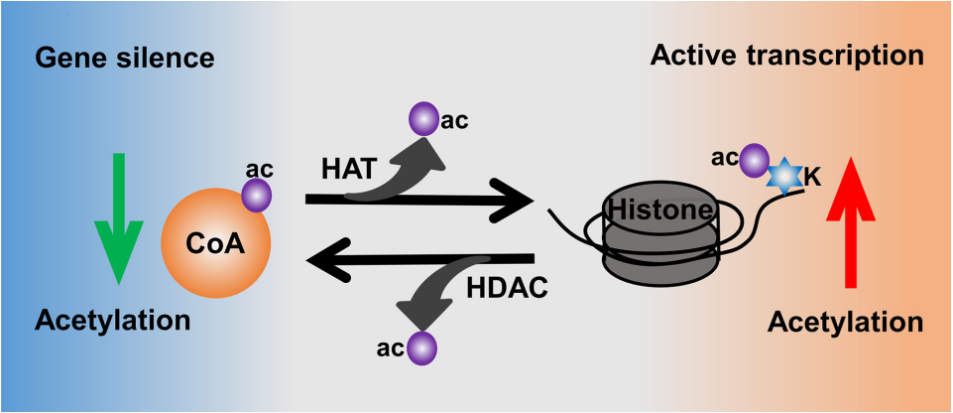
图2、HATs和HDACs介导组蛋白乙酰化[4]
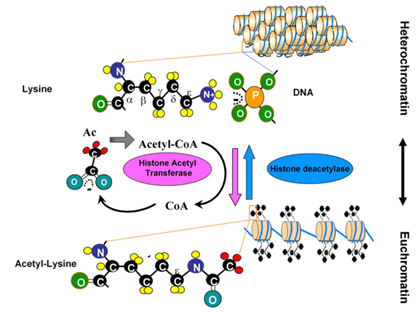
图3、组蛋白乙酰化和异染色质向常染色质的转变[6]
组蛋白乙酰化writer——HAT
根据细胞定位,HATs可分为A型和B型。A型HAT表现出核定位,B型HAT定位于细胞质中。
A型HAT催化染色质上的组蛋白发生乙酰化。与许多组蛋白修饰酶一样,A型HAT通常形成蛋白复合物,这些复合物中的组成蛋白在控制酶的补充、活性和底物特异性方面起着重要作用。例如纯化的酵母GCN5(具有乙酰转移酶活性)可乙酰化游离组蛋白,但不乙酰化核小体中的组蛋白,而当GCN5存在于SAGA复合物中时,能有效地乙酰化核小体组蛋白。根据同源性和乙酰化机制,A型HAT又分为5个家族:GNAT家族、CBP/p300家族、MYST家族、转录因子相关的HAT家族以及类固醇受体共激活因子。[2, 3]GNAT家族成员包括PCAF(KAT2B)、GCN5(KAT2A)和ELP3。PCAF和GCN5是哺乳动物中一对高度同源的HAT,除了具有组蛋白乙酰化酶活性外,还含有溴结构域(bromodomain),可以结合组蛋白H3、H4的乙酰化N端尾部,因此,它们既是乙酰化的“writer”又是“reader”[7]。CBP(KAT3A)和p300(KAT3B)是CBP/p300家族成员。CBP和p300在结构上高度同源,功能上冗余,但在实验鉴定的8707个启动子中,仍有222个启动子仅与p300相关,2747个启动子仅与CBP相关,有5738个启动子与两者都相关。CBP/P300家族的酶比其他HAT酶效率更高,底物特异性更低,被证明能乙酰化所有形式的组蛋白以及核小体。[6, 7]Tip60(KAT5)、MOZ(KAT6A)、MORF(KAT6B)、HBO1(KAT7)和MOF(KAT8)属于MYST家族。Tip60是一个进化保守复合物的催化亚基,可以乙酰化H2AK5、H3K14和H4K5、K8、K12、K16。MOF是MSL复合物的催化亚基,在果蝇中介导剂量补偿和调控基因表达。虽然人类MSL复合物的功能尚未完全明确,但研究发现,无论是果蝇还是人类MOF,都能特异性催化组蛋白H4K16的乙酰化。[7]转录因子相关的HAT家族包括TAFⅡ250(KAT4)和TIFⅢC90(KAT12)。TAFⅡ250是TFⅡD复合体中最大的亚基,除了具有乙酰转移酶活性外,还包含串联的溴结构域,在基因转录过程中发挥关键调控作用。[6]此外,几种类固醇受体共激活因子,如p600(KAT13C)、SRC1(KAT13A)、CLOCK(KAT13D)和NCOA3(KAT13B)等也属于A型HAT。[8]
B型HAT催化游离组蛋白乙酰化。这类酶在胞质中催化新合成组蛋白H4的K5和K12位点以及H3某些位点的乙酰化。这种独特的乙酰化模式对组蛋白的正确沉积至关重要,随后这些标记会被去除。B型HAT具有高度保守性,所有成员均与酵母Hat1序列同源。目前已知的B型HATs包括HAT1(KAT1)和HAT4(NAA60)。[3]
组蛋白乙酰化reader——溴结构域
组蛋白的乙酰化修饰不仅能够促进染色质相关蛋白的接近,还能为含有特定识别基序(即"reader")的蛋白质提供结合位点。其中,溴结构域(bromodomain,BRD)作为唯一能特异性识别乙酰化赖氨酸的相互作用模块,在这一过程中发挥着关键作用。BRD与乙酰赖氨酸的相互作用具有动态性,这与赖氨酸乙酰化的可逆特性相一致[9]。BRD是一个约110个氨基酸的进化保守模块,其典型结构为左旋四螺旋束(αZ、αA、αB、αC),螺旋束之间的ZA和BC环形成一个疏水口袋,既能维持结构的稳定性,又能特异性结合乙酰赖氨酸。虽然所有BRD的结合口袋都具有疏水性,但不同BRD家族成员在口袋开口处的静电相互作用存在显著差异。这种结构差异不仅决定了单个BRD的特异性,也为BRD蛋白的家族细分和特异性小分子药物的开发提供了可能。尽管BRD蛋白可能通过保守的结构折叠域与靶蛋白相互作用,但其对组蛋白的结合特异性主要源于整体序列的差异性,尤其在ZA环和BC环区域存在显著差异。有趣的是,参与乙酰赖氨酸识别的关键氨基酸残基(如PCAF中的Tyr760、Tyr802和Asn803)却在BRD家族中高度保守。[10]
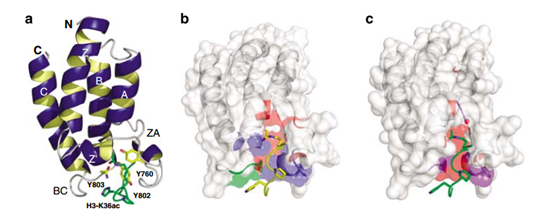
图4、BRD的结构和乙酰赖氨酸识别[10]
人类基因组编码的46种BRD相关蛋白共包含61个溴结构域,存在于许多不同的转录辅助调节因子和染色质修饰酶中,例如HAT和HAT相关蛋白、ATP依赖性染色质重塑复合物(BAZ1B)、解旋酶(SMARCA)等。根据其结构特征BRD可划分为8个不同的亚家族(Ⅰ~Ⅷ家族)[11]。
第Ⅰ亚家族包括PCAF、GCN5L2、FALZ、CECR2,这些蛋白都定位于细胞核中。PCAF的BRD在体外与乙酰化组蛋白H3(K9ac、K14ac、K36ac)以及组蛋白H4(K8ac、K16ac、K20ac)位点相互作用。事实上,正是PCAF的BRD证明了BRD结合乙酰化赖氨酸的能力。GCN5L2的BRD与组蛋白H2A (K5ac)、组蛋白H3 (K9ac、K14ac、K9ac/K14ac)和组蛋白H4 (K8ac/K14ac、K16ac、K5ac/K8ac/K12ac/K16ac)相互作用;FALZ与组蛋白H4 (K5ac)相互作用;CCECR2对组蛋白H3 (K9ac、K14ac)也有亲和力。
溴结构域和超末端结构域(Bromodomain and extraterminal domain, BET)蛋白家族属于第Ⅱ亚家族,该家族研究最为深入和广泛,其高度特异性小分子抑制剂已经显示出广阔的转化医学应用前景。BET家族包括BRD2、BRD3、BRD4和BRDT,它们具有相似的结构:N端有两个BRD,C端含有一个额外末端(ET)结构域。它们的两个BRD对具有单个或多个乙酰化标记的组蛋白H4都表现出亲和力。
第Ⅲ亚家族包含BRD8B、CREBBP、EP300、WDR9(2)、BAZ1B、BRWD3(2)和PHIP(2)。CREBBP和EP300的BRD与组蛋白的结合被广泛研究,CREBBP结合组蛋白H2B (K85ac)、组蛋白H3 (K9ac/K14ac、K14ac、K36ac、K56ac)以及组蛋白H4 (K12ac、K20ac、K44ac);EP300的BRD结合组蛋白H3 (K36ac,K56ac)和组蛋白H4 (K12ac,K20ac,K44ac)。
亚家族Ⅳ包含BRD7、BRD1、BRPF1、ATAD2、KIAA1240、BRD9和BRPF3。该家族的BRDs与乙酰化蛋白的结合机制尚未系统阐明。免疫共沉淀技术中ATAD2的BRD可特异性捕获组蛋白H3 (K14ac);而同源的KIAA1240对组蛋白H4 (K5ac)具有结合偏好性;BRD7的BRD与组蛋白H3 (K9ac,K14ac)以及组蛋白H4 (K8ac,K12ac,K16ac)存在低亲和力结合。
亚家族包含TRIM66、TRIM33、TIF1α、SP100、SP110、SP140、LOC93349、BAZ2A和BAZ2B。该家族的结构特征在于同时含有甲基化赖氨酸识别结构域(PHD finger)和BRD结构域,这种串联表观遗传阅读模块是实现与染色质结合的必要条件,同时揭示了染色质生物学的一个重要规律:含有多个reader结构域的表观遗传reader蛋白可通过多价协同作用识别组蛋白修饰。TIF1α通过PHD/BRD串联模块结合单乙酰化修饰的组蛋白H3 (K23ac)和H4 (K16ac);TRIM33A的PHD/BRD串联模块以亚微摩尔亲和力与单乙酰化和多乙酰化的组蛋白H3结合;BAZ2B的BRD对组蛋白H3 (K14ac)具有个位数的微摩尔亲和力。
亚家族包含MLL和TRIM28,目前尚未见关于该亚家族与组蛋白或其他蛋白质通过乙酰化赖氨酸相互作用的报道。
亚家族Ⅶ包含ZMYND11、TAF1、TAF1L、WDR9(1)、BRWD3(1)和PHIP(1)。TAF1通过BRD结构域以低微摩尔亲和力协同结合组蛋白H4上的多个Kac标记(K8ac/K16ac, K5ac/K12ac, K5ac/K8ac/K12ac/K16ac),当然TAF1也与单个Kac标记的组蛋白H4结合(K8ac、K12ac、K16ac),与组蛋白H3(K14 ac,K9 ac/K14 ac)的结合也已得到了证实。
最后一个亚家族(Ⅷ家族)包含ASH1L、SMARCA2、SMARCA4以及PB1。SMARCA2的BRD已被证实与组蛋白H3(K9ac、K14ac和K9ac/K14ac)以及组蛋白H4(K8ac、K12ac、K16ac和K5ac/K8ac/K12ac/K16ac)结合;据报道SMARCA4结合组蛋白H2B(K5ac)、组蛋白H3(K14ac、K9ac/K14ac)以及组蛋白H4(K8ac和K12ac、K16ac);PB1蛋白的BRD也显示出结合组蛋白H3 N端赖氨酸标记(K4ac、K9ac、K14ac、K18ac、K23ac)。
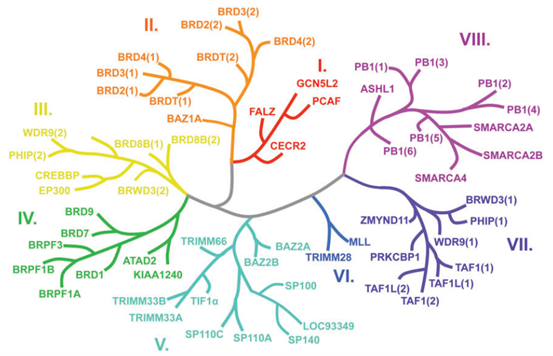
图5、基于人类BRDs结构的系统进化树[9]
组蛋白乙酰化eraser——HDAC
执行乙酰化逆反应(即脱乙酰化)是HDAC的核心功能。根据与酵母脱乙酰酶的序列相似性,HDAC可分为2个家族。第一个家族成员需要Zn2+作为酶活性的辅助因子,包括I类(HDAC1、2、3和8)、Ⅱ类(HDAC4、5、6、7、9和10)和Ⅳ类(HDAC11),其中Ⅱ类酶根据结构域组成又分为Ⅱa类(HDAC4、5、7、9)和Ⅱb类(HDAC6、10)。脱乙酰酶的第二个家族即Ⅲ类HDAC,需要NAD+作为辅助因子,其成员最广为人知的是sirtuin蛋白。尽管HDAC本身的底物特异性相对较低,每种酶都能催化多个不同的组蛋白位点脱乙酰化,但它们通常存在于多种不同的复合物中,从而在细胞内实现功能特异性。[3, 8, 12]
执行乙酰化逆反应(即脱乙酰化)是HDAC的核心功能。根据与酵母脱乙酰酶的序列相似性,HDAC可分为2个家族。第一个家族成员需要Zn2+作为酶活性的辅助因子,包括I类(HDAC1、2、3和8)、Ⅱ类(HDAC4、5、6、7、9和10)和Ⅳ类(HDAC11),其中Ⅱ类酶根据结构域组成又分为Ⅱa类(HDAC4、5、7、9)和Ⅱb类(HDAC6、10)。脱乙酰酶的第二个家族即Ⅲ类HDAC,需要NAD+作为辅助因子,其成员最广为人知的是sirtuin蛋白。尽管HDAC本身的底物特异性相对较低,每种酶都能催化多个不同的组蛋白位点脱乙酰化,但它们通常存在于多种不同的复合物中,从而在细胞内实现功能特异性。[3, 8, 12]
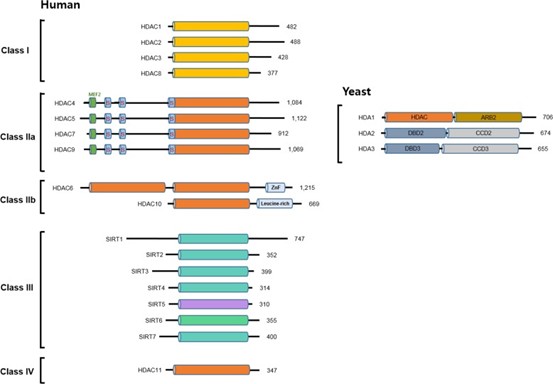
图6、人和酵母HDAC的分类和结构域组成[13]
I类HDAC与酵母Rpd3高度同源,由一个完全保守的脱乙酰酶结构域组成,普遍定位于细胞核中,对组蛋白表现出很强的脱乙酰酶活性。其中HDAC8比较特殊,它不形成大型复合物,而是单独发挥作用。
Ⅱ类HDAC与酵母HDA1高度同源,其核心特征为C端含有保守的脱乙酰酶催化结构域,这些酶通常存在于细胞质中。Ⅱa类HDAC包含一个独特的N端接头结构域,该结构域具有MEF2的结合位点和多个磷酸化位点,这些修饰位点可以被14-3-3蛋白识别。这种特殊的结构组成使Ⅱa类HDAC能够响应胞外信号在核质间动态穿梭。尽管具有完整的催化结构域,Ⅱa类HDAC却表现非常低的酶活性,暗示其可能作为低酶活性的脱乙酰酶发挥作用,或者可能存在尚未鉴定的特定靶标。相比之下,Ⅱb类HDAC的C端结构有显著不同,HDAC6包含两个脱乙酰酶结构域和一个C端锌指泛素结合结构域;而HDAC10在C端只有一个脱乙酰酶结构域和一个富含亮氨酸的重复结构域。目前对Ⅱb类HDAC的生理功能和调控机制还知之甚少。
Ⅳ类HDAC仅包括HDAC11,它与酵母Hos3同源,与DNA复制因子CDT1和IL10表达有关。
Ⅲ类HDAC与酵母Sir2同源,这是从细菌到人类的许多生物中广泛保守的蛋白质。脱氧hypusine合酶样NAD/FAD结合域是Ⅲ类HDAC的一个显著特征。在人类中已经报道了7种sir2样蛋白(SIRT1-7),它们被称为sirtuins。Sirtuins可能存在于细胞核(SIRT1、SIRT2、SIRT3、SIRT6和SIRT7)、细胞质(SIRT1和SIRT2)或线粒体(SIRT3、SIRT4和SIRT5)中。这些sirtuins的一个独特特征是它们表现出其他酶活性,例如SIRT5具有赖氨酸脱琥珀酰化酶和脱丙烯酰化酶活性。
长期以来,表观遗传学领域普遍认为HDAC与被抑制的基因结合,并在基因激活时被HAT取代。然而,Wang等人的数据揭示了颠覆性的发现:所有被检测的HDAC(包括HDAC1-3、HDAC6)都富集于转录活跃的基因,而非沉默基因。并且所有测试的HDAC均与mRNA表达水平和Pol Ⅱ水平正相关。有趣的是,HDAC似乎也与组蛋白乙酰化水平正相关。进一步统计发现,人类基因组中的大多数HDAC与活性基因相关,只有一小部分在沉默基因中检测到。这些数据表明HDAC的一个主要功能是去除活性基因中HAT添加的乙酰基,并在基因激活后重置染色质修饰。[7]
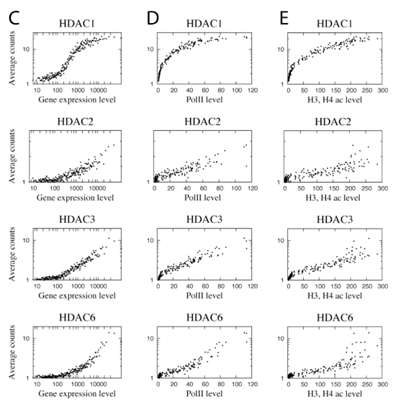
图7、HDAC与基因表达、Pol Ⅱ结合和组蛋白乙酰化水平相关性[7]
组蛋白乙酰化在细胞生理和疾病中的作用
组蛋白乙酰化作为一种重要的表观遗传修饰,在生物体发育、疾病发生和治疗中发挥着关键作用。不同的组蛋白乙酰化修饰在发育过程中的功能已经在许多生物体中得到证实:例如,在鸡成肌细胞分化过程中,HAT活性呈现时间依赖性变化;在大鼠骨骼肌中,染色体蛋白的乙酰化随着发育的进展而减少;在真兽亚纲动物中,第二个X染色体失活发生在雌性后代的细胞中,虽然这种失活的机制尚未明确,但组蛋白乙酰化和X染色体失活之间存在令人信服的联系;此外,在X染色体拷贝数异常的患者中,观察到所有X染色体的不完全脱乙酰化。基因突变研究进一步证实了HDACs的关键作用:HDAC1缺失的小鼠胚胎在第10.5天前死亡并出现严重的增殖缺陷和生长迟缓;HDAC3突变不仅导致小鼠在胚胎期第9.5天前死亡,还和DNA双链断裂修复系统缺陷有关;而HDAC7缺失则通过影响血管生成导致小鼠胚胎死亡。[6]
组蛋白翻译后修饰与炎症性疾病的发生同样密切相关。在动脉粥样硬化中,lncRNA NORAD通过募集HDAC6促进H3K9脱乙酰化,抑制VEGF表达,从而减少血管内皮损伤;Ren等人对哮喘患者肺组织的蛋白质组学分析发现了15个差异修饰的乙酰化位点,其中13个位点上调(包括H3K9ac、H3K14ac、H3K18ac、H3K23ac、H3K27ac、H3K36ac、H2B1K120ac、H2BK20ac、H2BK16ac、H2BK20ac、H2BK108ac、H2BK116ac和H2BK120ac),2个位点下调(包括H2BK5ac和H2BK11ac);SV40病毒目前已经成为病毒感染后组蛋白修饰的模型系统,病毒DNA和组蛋白形成的微小染色体中存在H3和H4超乙酰化现象。[4]
当组蛋白乙酰化调控失衡时,可能导致恶性肿瘤的发生。基因表达紊乱作为癌症的重要标志,与HAT功能异常密切相关,在多种癌症中发现了p300和CBP基因单个等位基因的体细胞突变;缺失CBP或P300的转基因小鼠会罹患血液系统恶性肿瘤;HAT也可以通过染色体易位与不同的融合基因发生融合而获得致癌特性(如MLL-CBP、MLL-EP300、MOZ-EP300或MOZ-CBP),这可能是因为HATs对其融合伙伴的基因组靶点进行异常乙酰化[14]。与HAT协同调控表观遗传的HDAC被发现与两种重要的细胞周期调节因子Mad/Max和RB有关,Mad/Max的转录抑制需要装配一个携带HDAC活性的多亚基阻遏复合体。这一机制揭示了HDACs在细胞周期调控中的枢纽地位,表明其可能参与肿瘤的发生[6]。
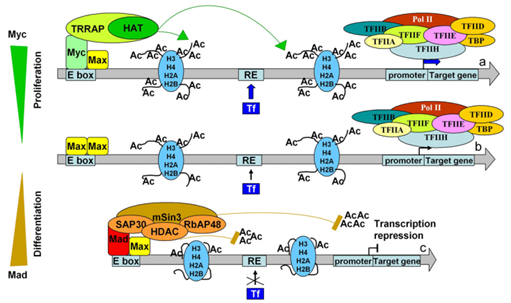
图8、组蛋白乙酰化在细胞发育调控中的作用的模型[6]
基于此,靶向HDAC催化结构域成为抗癌药物研发的重要方向。目前已开发出SAHA(vorinostat)和FK288(romidepsin)等竞争性抑制剂,并获FDA批准用于抗癌治疗。然而,与多数抗癌药物类似,HDAC抑制剂仍面临疗效局限、耐药性及剂量依赖性毒性等挑战[13]。HAT作为表观遗传调控的关键酶被视为潜力靶点,但其抑制剂的研发进程显著落后于HDAC抑制剂[14]。这一困境促使研究者探索替代策略,例如靶向BET家族蛋白。目前已开发出I-BET762、JQ1和I-BET151等高特异性BET BRD抑制剂,这类药物具有高效细胞渗透性,能竞争性取代乙酰化组蛋白上的BET蛋白,从而阻断致癌转录,为克服传统HDAC/HAT靶向治疗的局限性提供了新方向[9]。
然而,乙酰化修饰研究的发展并非一帆风顺。自1964年组蛋白乙酰化现象首次被发现后[15],这一领域在近30年间长期处于边缘地位。直到1996年,Allis和Schreiber团队发表了两篇论文,系统揭示了组蛋白乙酰化在基因调控中的核心作用,才真正引起学术界关注,相关论文年发表量呈现爆发式增长。
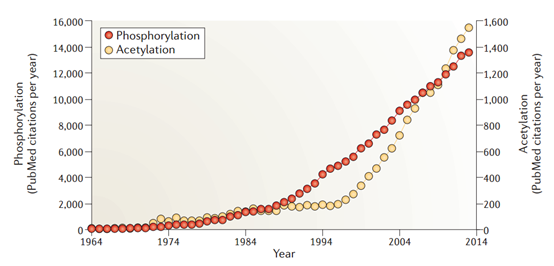
图9、关于蛋白质乙酰化的出版物[16]
如今,组蛋白修饰调控机制已被证实参与细胞分化、免疫应答、代谢稳定等几乎所有生物过程,尤其是乙酰化机制在肿瘤、神经退行性疾病中的关键作用,使其成为表观遗传药物研发的焦点。但是这些成果的转化应用仍依赖于对乙酰化动态调控网络的深度解析。汉恒生物专营病毒包装十余载,建立了庞大的基因研究现货工具库,可为广大研究人员提供多种乙酰化修饰相关基因的病毒现货。若有其他病毒包装需求或疑问,也欢迎随时咨询汉恒生物微信公众号或拨打官网技术服务热线:400-092-0065。
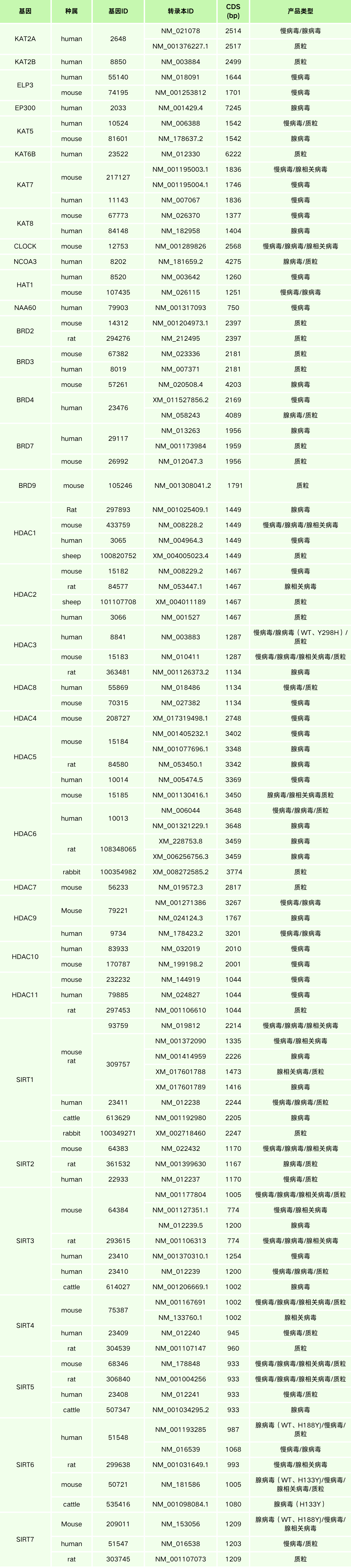
汉恒生物组蛋白乙酰化相关基因现货
参考文献
1. Liu C, Lu F, Cui X, Cao X. Histone methylation in higher plants. Annu Rev Plant Biol 2010, 61: 395-420
2. Sun XJ, Man N, Tan Y, Nimer SD, Wang L. The Role of Histone Acetyltransferases in Normal and Malignant Hematopoiesis. Front Oncol 2015, 5: 108
3. Bannister AJ, Kouzarides T. Regulation of chromatin by histone modifications. Cell Res 2011, 21: 381-395
4. Lin Y, Qiu T, Wei G, Que Y, Wang W, Kong Y, Xie T, et al. Role of Histone Post-Translational Modifications in Inflammatory Diseases. Front Immunol 2022, 13: 852272
5. Mihaylova MM, Shaw RJ. Metabolic reprogramming by class I and II histone deacetylases. Trends Endocrinol Metab 2013, 24: 48-57
6. Khan SN, Khan AU. Role of histone acetylation in cell physiology and diseases: An update. Clin Chim Acta 2010, 411: 1401-1411
7. Wang Z, Zang C, Cui K, Schones DE, Barski A, Peng W, Zhao K. Genome-wide mapping of HATs and HDACs reveals distinct functions in active and inactive genes. Cell 2009, 138: 1019-1031
8. Alaskhar Alhamwe B, Khalaila R, Wolf J, von Bülow V, Harb H, Alhamdan F, Hii CS, et al. Histone modifications and their role in epigenetics of atopy and allergic diseases. Allergy Asthma Clin Immunol 2018, 14: 39
9. Barbieri I, Cannizzaro E, Dawson MA. Bromodomains as therapeutic targets in cancer. Brief Funct Genomics 2013, 12: 219-230
10. Mujtaba S, Zeng L, Zhou MM. Structure and acetyl-lysine recognition of the bromodomain. Oncogene 2007, 26: 5521-5527
11. Filippakopoulos P, Knapp S. The bromodomain interaction module. FEBS Lett 2012, 586: 2692-2704
12. Araki Y, Mimura T. The Histone Modification Code in the Pathogenesis of Autoimmune Diseases. Mediators Inflamm 2017, 2017: 2608605
13. Park SY, Kim JS. A short guide to histone deacetylases including recent progress on class II enzymes. Exp Mol Med 2020, 52: 204-212
14. Audia JE, Campbell RM. Histone Modifications and Cancer. Cold Spring Harb Perspect Biol 2016, 8: a019521
15. Allfrey VG, Faulkner R, Mirsky AE. ACETYLATION AND METHYLATION OF HISTONES AND THEIR POSSIBLE ROLE IN THE REGULATION OF RNA SYNTHESIS. Proc Natl Acad Sci U S A 1964, 51: 786-794
16. Verdin E, Ott M. 50 years of protein acetylation: from gene regulation to epigenetics, metabolism and beyond. Nat Rev Mol Cell Biol 2015, 16: 258-264

查看更多

查看更多

查看更多

联系我们

返回顶部